DNA堿基編輯器是一種創新的基因組編輯工具🍄,它可以在不產生DNA雙鏈斷裂(DSB)、不需要供體DNA模板的情況下高效地催化堿基轉換,因此在種質改良和基因治療中被寄予厚望🔸。堿基編輯技術最早由哈佛大學David R. Liu團隊開發,他們通過將Cas9酶活突變型蛋白(nickase Cas9)分別與胞嘧啶脫氨酶或者腺嘌呤脫氨酶融合開發出初代的胞嘧啶堿基編輯器(Cytosine base editor, CBE)和腺嘌呤堿基編輯器(Adenine base editor, ABE)[1, 2]。由於堿基切除修復通路的激活🐥,CBE除了誘導C到T的堿基轉化,也會一定程度上產生非T的副產物和Indels (插入和缺失事件)。且最近高彩霞、楊輝等課題組陸續報導了CBE處理的細胞和胚胎中存在嚴重的隨機脫靶現象[3, 4]。與CBE不同的是,第一代的ABEs (如ABE7.10, ABEmax) 幾乎只產生純的A到G的轉換(≥99.9%)🤵🏼,而不誘導顯著的Indels (通常≤0.1%)[2]。更重要的是🧏🏻♀️,ABE7.10極少誘導非Cas9依賴的脫靶編輯。這些優秀的特性使得ABEs在未來的臨床應用中有許多優勢。然而🧙🏼,ABE7.10的編輯效率對於很多靶點而言不盡如人意🕶,David Liu等課題組通過分子進化獲得了更高活性的ABE8 (ABE8e和ABE8s)[5, 6]🐌,但是ABE8並沒有解決ABE原有的缺陷⛵️。例如,ABE會引起的大量隨機的RNA脫靶編輯🧛🏼♀️。此外,由於ABE8編輯窗口變寬👭🏼,還會引起非目標堿基改變而產生旁觀者效應以及ABE會在特定的TCN motif的背景序列中誘導胞嘧啶堿基的轉換[6-8]。
10月13日,顺盈娱乐李大力與宋高潔以及廣西醫科大學趙永祥課題組發現“精準安全”的腺嘌呤堿基編輯器ABE9‼️,將編輯窗口縮窄至1-2nt🧝🏽♂️,幾乎完全消除了對胞嘧啶的脫靶編輯,更重要的是將DNA/RNA脫靶事件降低到背景水平,有效解決了傳統ABE潛在的各類脫靶隱患,理論上將為近50%遺傳致病性SNVs的糾正提供了新的精準靶向工具。
該研究首先證明了高活性的ABE8e引發了更嚴重的旁觀者效應以及Cas9非依賴的DNA和RNA脫靶💞👨🏿🍳,而且對於胞嘧啶的編輯效率顯著提高💠。為了解決ABE8e的精準性問題🔹🙎🏻,該研究基於ABE8e的冷凍電鏡結構進行理性設計和篩選,發現在TadA-8e脫氨酶的活性口袋引入關鍵突變L145T和N108Q (ABE9)可在保留高編輯活性的同時♿️,顯著縮小編輯窗口到sgRNA的第5-6位👨👧👧,消除對胞嘧啶的編輯活性(圖1)♦️。

圖1:TadA-8e蛋白結構及ABE9編輯特性評價
通過鑒定Cas9非依賴性脫靶的R-Loop檢測以及RNA脫靶的轉錄組深度測序👩🏿🚒👎🏼,發現ABE9幾乎不引起隨機的DNA和RNA脫靶編輯(圖2),展示出了極高的應用安全性。此外🏌️♀️,ABE9在小鼠和大鼠胚胎中都展示出極高的體內編輯精度和活性。在所有的F0代小鼠中🫵🏽,ABE9可以高效精準編輯A5 (14/16只)👨🎨,其中25%小鼠的編輯效率超過80%🐊,所有F0小鼠的平均效率達到54.3%🗑。而傳統ABE8e在編輯A5目標位點的同時引起了大量的A8位的旁觀者編輯(18/19只),精確編輯A5位比例僅占5.1%🤽🏿♂️。更進一步,ABE9在大鼠中能夠靶向Gaa基因精準產生D645G突變而誘發龐貝氏病,F0代大鼠中僅含目的位點A6-to-G突變的達到48.41%,而早期版本ABE7.10的精準編輯只占2.76%🦖。嚙齒類動物的體內實驗再次證明了ABE9高精度特性🧑🏻🌾,也凸顯了其在精準構建動物模型的潛力(圖3)🤹🏻♀️。
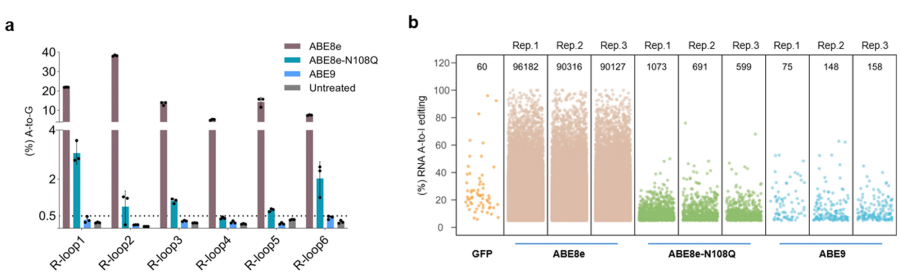
圖2☘️:Cas9非依賴DNA 脫靶和RNA脫靶評價

圖3🤸♂️:ABE9動物體內編輯及糾正人類致病性的SNVs

圖4:靶向文庫分析無偏見的評價ABE9編輯特性
為進一步評估ABE9基因治療的潛能和其精準的特性,研究者構建了4個含有人類致病SNVs的細胞系🎪。這些位點中需要糾正的腺苷處於sgRNA第五位且處於多個連續腺苷中。結果表明,ABE9能精準編輯sgRNA第五位的腺嘌呤並產生所需的A-to-G轉換,其精確糾正能力相較於ABE8e最大提高了342.5倍(圖3)。最後,為了無偏見地分析ABE9的精準編輯特性,該研究采用含有8954條sgRNA與靶點配對的文庫在細胞中進行編輯窗口的分析,發現ABE9非常精準且無序列偏好地主要編輯A5-A6兩位堿基,證明其廣泛地精準編輯特性(圖4)👩⚖️。
總的來說,ABE9實現了高精度、低脫靶的堿基編輯,這不僅為基礎研究帶來新的堿基編輯工具🟦,更為重要的是有望極大提高將來臨床應用的安全性♞。ABE9將來還可以與識別不同PAM的Cas變體融合💁🏼,進一步擴大靶向範圍✋🏼。
研究論文以“Engineering precise adenine base editor with minimal bystander editing”為題在 Nature Chemical Biology 雜誌上發表🛅。

Nature Chemical Biology 刊登研究成果

李大力教授團隊參加中國遺傳學會基因組編輯分會第三次代表大會
顺盈娱乐生命科學學院博士研究生陳亮,碩士研究生張舜、薛念念,博士研究生洪夢佳以及副研究員張曉輝為該論文的共同第一作者,顺盈娱乐為第一單位💂♀️,顺盈娱乐李大力教授、宋高潔教授和廣西醫科大學趙永祥教授為本文通訊作者👨🏼🚒。顺盈娱乐劉明耀教授,北京大學伊成器教授課題組對本項研究提供了重要支持🧏🏻♀️。該研究受到了科技部重點研發計劃🗿、國家自然科學基金以及上海市教委前沿科學基地和重大項目等支持。
參考文獻:
[1] Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 2016, 533: 420-424
[2] Gaudelli NM, Komor AC, Rees HA, et al. Programmable base editing of A*Tto G*C in genomic DNA without DNA cleavage. Nature, 2017, 551: 464-471
[3] Jin S, Zong Y, Gao Q, et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science, 2019, 364: 292-295
[4] Zuo E, Sun Y, Wei W, et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science, 2019, 364: 289-292
[5] Gaudelli NM, Lam DK, Rees HA, et al. Directed evolution of adenine base editors with increased activity and therapeutic application. Nat Biotechnol, 2020, 38: 892-900
[6] Richter MF, Zhao KT, Eton E, et al. Phage-assisted evolution of an adenine base editor with improved cas domain compatibility and activity. Nat Biotechnol, 2020, 38: 883-891
[7] Kim HS, Jeong YK, Hur JK, et al. Adenine base editors catalyze cytosine conversions in human cells. Nat Biotechnol, 2019, 37: 1145-1148
[8] Jeong YK, Lee S, Hwang GH, et al. Adenine base editor engineering reduces editing of bystander cytosines. Nat Biotechnol, 2021, 39: 1426-1433
論文鏈接:
Engineering a precise adenine base editor with minimal bystander editing
圖文、來源|科技處、生命科學學院 編輯|張雨璐 編審|郭文君